医学相关的图像分割技术
| 医学2D | 医学3D |
|---|---|
| A Novel Domain Adaptation Framework for Medical Image Segmentation | 3D U-Net: Learning Dense Volumetric Segmentation from Sparrse Annotation |
| DeepMedic for Brain Tumor Segmentation | Joint Sequence Learning and Cross-Modality Convolution for 3D Biomedical Segmentation |
| Simultaneous Super-Resolution and Cross-Modality Synthesis of 3D Medical Image using Weekly-Supervised Joint Convolutional Sparse Coding |
A Novel Domain Adaptation Framework for Medical Image Segmentation
脑部肿瘤切割
- 难点在于难以精确定位肿瘤(肿瘤形状各异,分布广泛)
- 核磁共振4种模态(Modality)
- 自旋晶格驰像(T1)
- T1对比(T2)
- 自旋松弛(T2)
- 流体衰减反转恢复(FLAIR)
论文创新点:
- A biophysics based domain adaptation method(物理肿瘤生长模型加上对抗网络生成逼真的MR图像补充数据集)
- An automatic method to 分割健康的组织(白质灰质,脑脊液),通过健康的组织轮廓辅助脑补图像切割
方法
- Data Augmentation: 使用基于生物学的肿瘤生长模型(PDE)模拟合成的肿瘤,在使用一个辅助的神经网络修正模拟肿瘤到correct intensities distribution对比真实的MR图像(会通过强加循环一致性限制分布情况)
Extened segmentation: 扩展分割到健康的薄壁组织。先使用(in-house diffeomorphic registration code)微分同胚技术(是一种光滑可逆的变换,在MRI图像配准中可以保证形变后的拓扑结构保持不变,同时避免出现不合理的物理现象)处理过的数据进行训练。
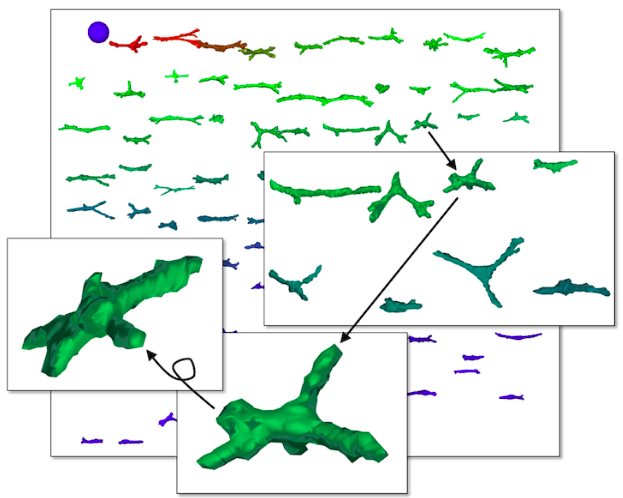
然后再使用DNN去分割健康的组织(神经胶质,脑脊液,灰质和白质),结果如上图。 这样可以增加健康组织的轮廓作为重要的训练信息并且改进了原来的类不平衡的问题。
具体的步骤分为:
- Affine registration of each atlas image to the brats image
- Diffeomorphic registration of each atlas image to the BraTS image(把健康组织的信息匹配BraTS的数据集结构)
- Majority voting to fuse labels of all deformed atlases to get the final healthy tissuse segmentation
模型
3D U-Net(会在下面讨论)
使用3D进行第一阶段的检测,获得肿瘤的初始位置。
U-Net(在前一章节讲过)
使用2D的U-Net以及domain adaptation results获得最终的分割结果
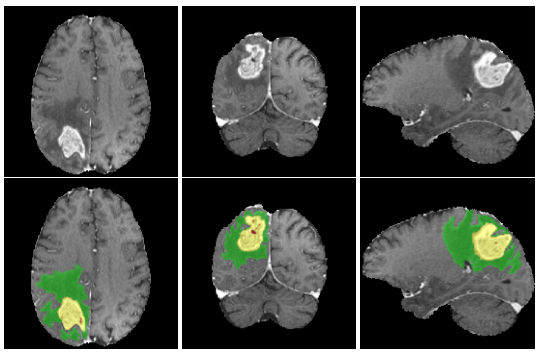
缺点
这个框架只支持2D的domain trasformations,所以对3D的数据只能进行切片并且最后是用的2D的神经网络,这样没有3D的网络来的efficient以及精确。
3D U-Net: Learning Dense Volumetric Segmentation from Sparse Annotation
在生物医学领域,3D数据是很多的,一层一层转化为2D数据去标注训是不现实的(及其耗时),而且用整个3D体积的全部数据去训练既低效又极有可能过拟合(相邻切片的数据是非常相近的)。这篇文章提出的3D Unet只需要少部分2D的标注切片就可以生成密集的立体的分割。此网络主要有两个不同的作用方式:
- Semi-automated setup: 在一个少量稀疏标注的数据集上训练并在此数据集的图像上预测其他未标注的地方。
- Fully-automated setup: 在representative的稀疏标注的数据集训练,然后用来切割新的图像。
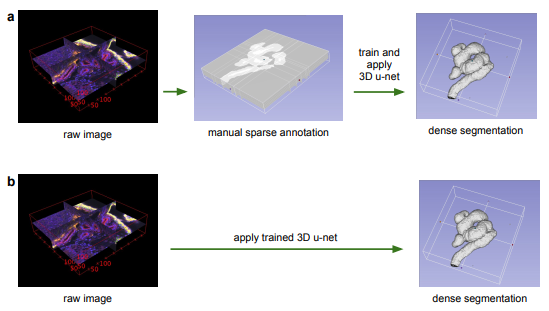
改进
- 在2D U-Net的基础上,仍然使用encoder去分析整个图片, 但是扩展了decoder来产生full-resolution的切割。
- 使用3D数据作为输入,因此网络改用3D convolutions, 3D max pooling 和 3D up-convolutional。
- 避免使用bottlenecks结构因为输入的数据不会很多,避免丢失重要的信息。
网络
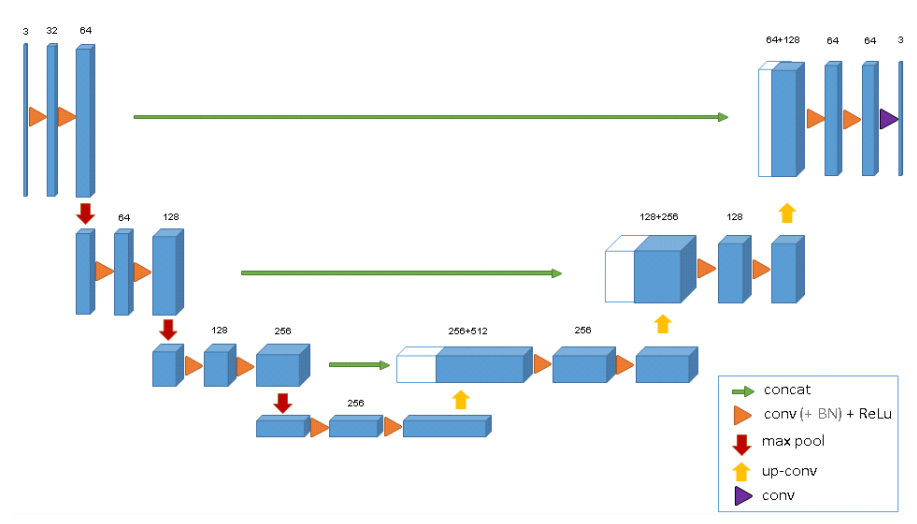
- Encoder部分: 每层包含两个$3 \times 3 \times 3$卷积和ReLU, 然后是一个strides为2的$2 \times 2 \times 2$的最大池化层。
- Decoder部分: 每层包含strides为2的$2 \times 2 \times 2$的upconvolution以及两个$3 \times 3 \times 3$卷积和ReLU
- 这里作者还用了Batch Normalization 防止梯度爆炸,并且在BN后增加了缩放和平移$x_{new}=\alpha \cdot x + \beta$, 其中两个超参是学习出来的。
- 能在稀疏标注的训练集训练的原因是使用了weighted softmanx loss function, 把unlabeled pixels的权重置为0让网络只学习有标注的部分并且提高了泛化能力。
Joint Sequence Learning and Cross-Modality Convolution for 3D Biomedical Segmentation
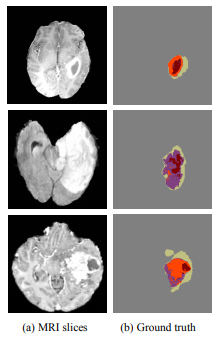
目前对于定位肿瘤的难点在于:
- 肿瘤形状各异,例如神经胶质瘤与胶质母细胞瘤形状不同
- 肿瘤分布广泛,可能分布于大脑的任何区域
这篇文章提出来一个思路就是交叉形态卷积的方法做一个 encoder-decoder 的网络结构,然后同时用LSTM对2D的切片序列建模。
MRI也是跟CT一样断层扫描的过程且包含4种模态(Modality),就是它一层一层,一层扫出来的就是一个2D的图片,然后多层累计起来就是3D的,但是其实切割是要切割出3D的脑部肿瘤位置,这样就需要把2D的变成3D的,把2D的切片之间的关系通过LSTM描述出来,最后把多模态卷积和LSTM网络结合在一起,达到3D切割。
模型
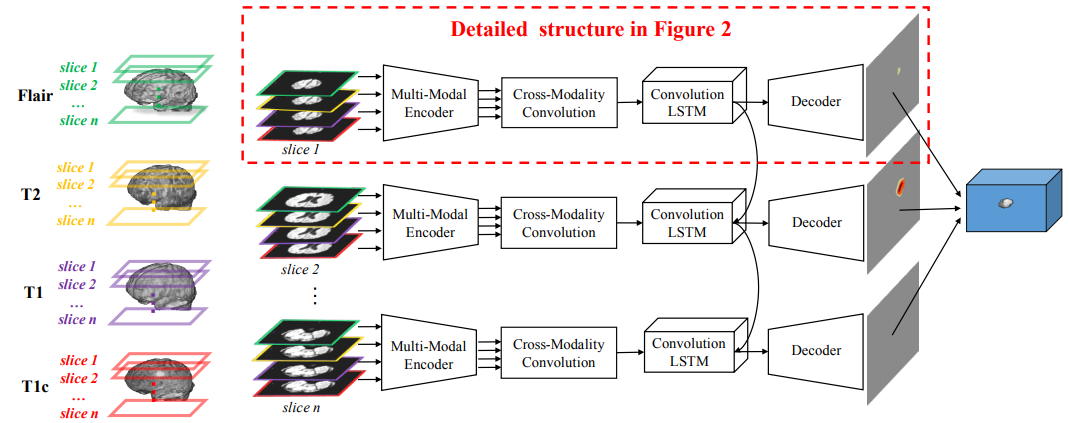
这个方法的framework大概是这样的,从左到右看。
首先每一个脑部的MRI数据,他都是通过四种模态切出来的,这里用四种不同的颜色来表示,相当于每一个slice就是我说的那个2D的图片。
切完之后他会把四个模态,就是到图b这个阶段了,四个模态交叉在一起做一个multi-modal的encoder,这个encoder就是用一个神经网络来实现的。
四个模态encode到一起之后,在这一步就用神经网络把四个模态下的脑部切割出来了,这是2D的情况下。
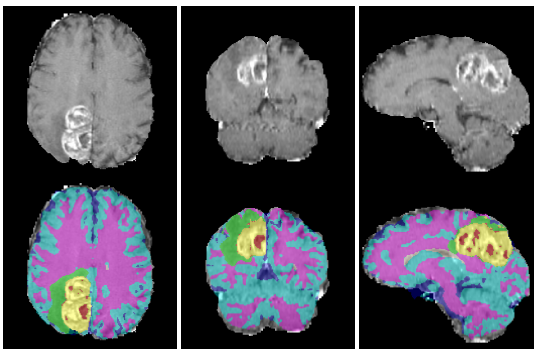
然后再加上convolution LSTM把2D的切割、2D和2D之间的dependency描述出来之后就形成了3D的切割,然后再做一下decoder,展现成最后这种形式。在最中间有一个切割出来的东西,其他没被切割到的background。
方法
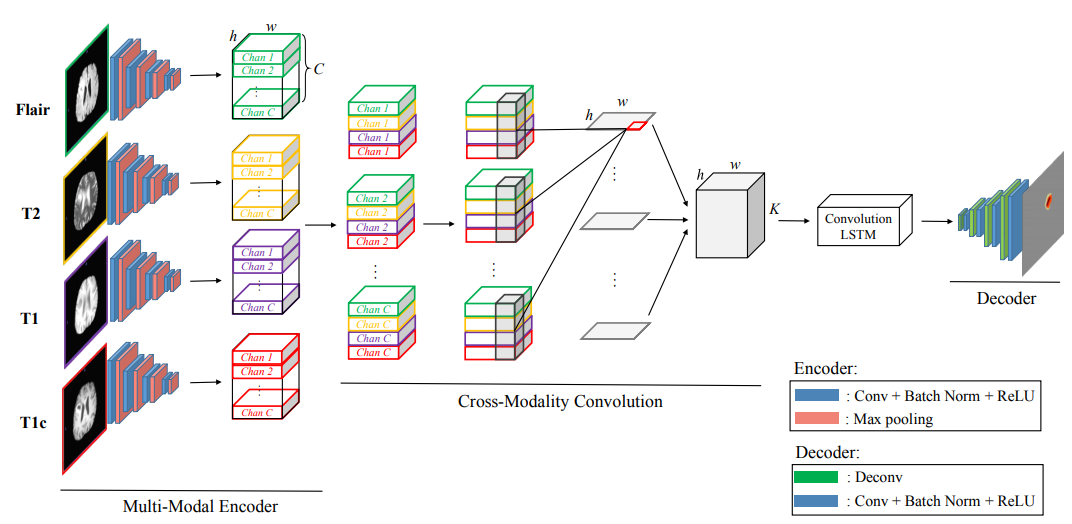
MME(Multi-Modal Encoder)
类似于SegNet里的编码器结构, 因为数据集比较小,因此网络也简化了,使用四个卷积核,通过batch-normalization,然后加一个非线性变换,在后面有四个最大池化层。
MRF(Multi-Resolution Fusion)
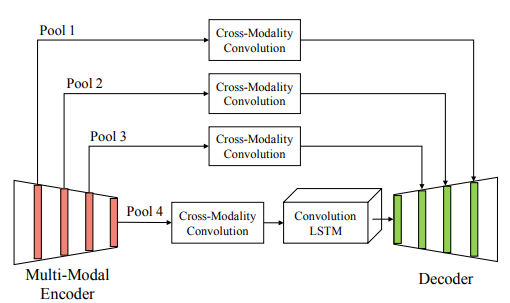
结合多尺度多模态的信息,通过在不同的尺度的encoder和decoder中进行feature multiplication代替级联因此不会增加特征映射的大小。CMC(Cross-Modality COnvolution)
CMC可以把空间信息以及不同模态的关系结合到一起。
四个模态的数据进入到这个卷积网络之后,就会把相同channel下的每一个模态stack在一起形成一个block, 就是每个channel里面有 C 个slice,就是说它是一个立体结构了,一个的长宽是H、W,高是C的这种。四个模态弄到一起就是C×4×H×W。然后通过一个三维的卷积,卷积的大小里有个C×4,也就是用4×1×1的一个卷积核,做卷积之后得到每一层的切割出来的特征。
Slice Sequence Learning
使用一个端到端的切片序列学习框架去建模切片之间的相关性。这个convolution LSTM跟普通的LSTM有一个区别,就是把原来的矩阵相乘替换为一个卷积操作,就是普通的乘法变成卷积层,这样它就能够在把之前状态的空间信息保留着。其实它的目的就是,卷积LSTM会描述一个2D切割边缘的趋势,比如说这一张中切片它的形态是这样的,然后到下一张它会有一个轻微的变化,要把这种变化描述出来。
此外不同切片的convLSTM的权重是共享的因此需要更新的参数不会随着序列增加。
Decoder
包含上采样以及soft-max进行分类
训练
Single Slice Training:
使用了median frequency balancing调节cross-entropy loss的权重来缓解数据不平衡的情况(98%的都是健康区域)
$$\alpha_{c}=\frac{median_freq}{freq(c)}$$Two-Phase Training:
- 第一阶段:只采样包含肿瘤的切片并且使用median frequency balancing的方法进行训练。
- 第二阶段:去除median frequency策略并且调低学习率,在真实的肿瘤分布概率下进行训练
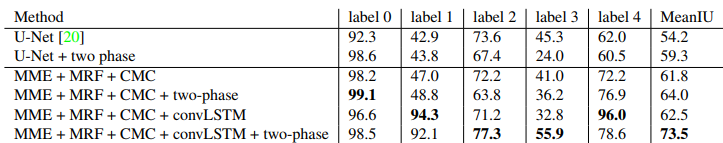
结果